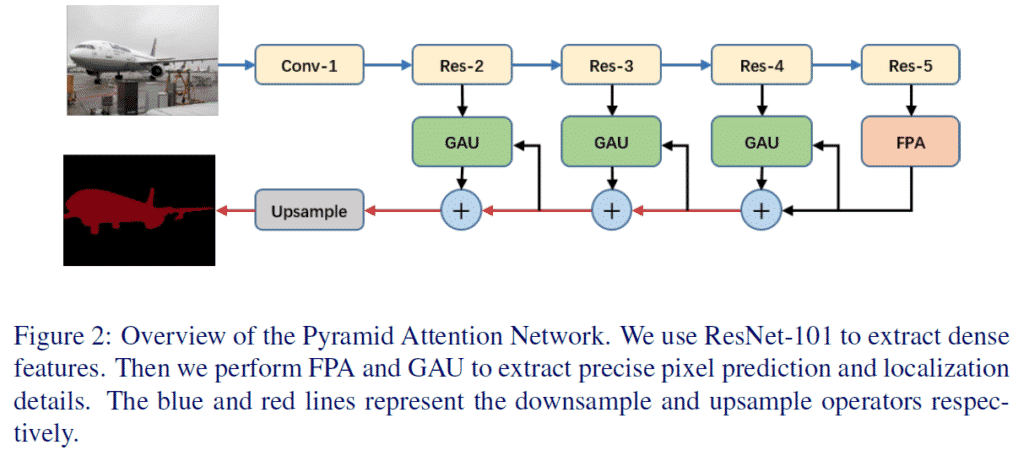
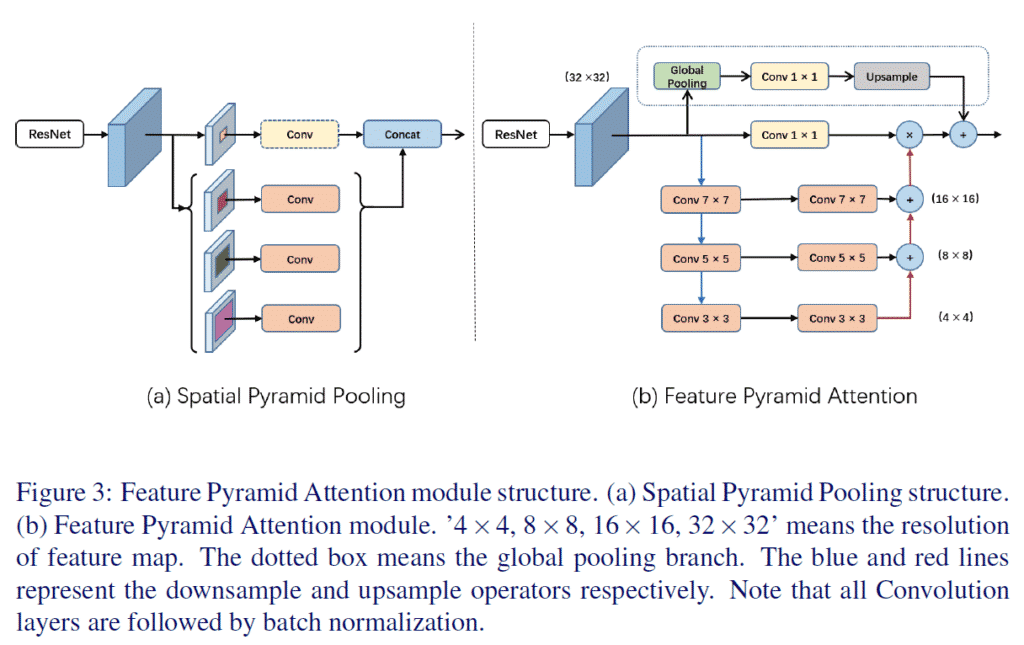
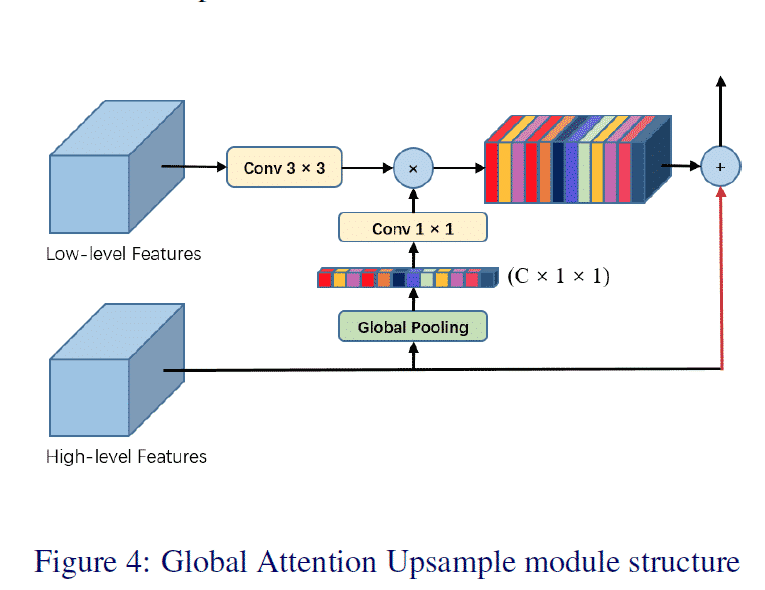
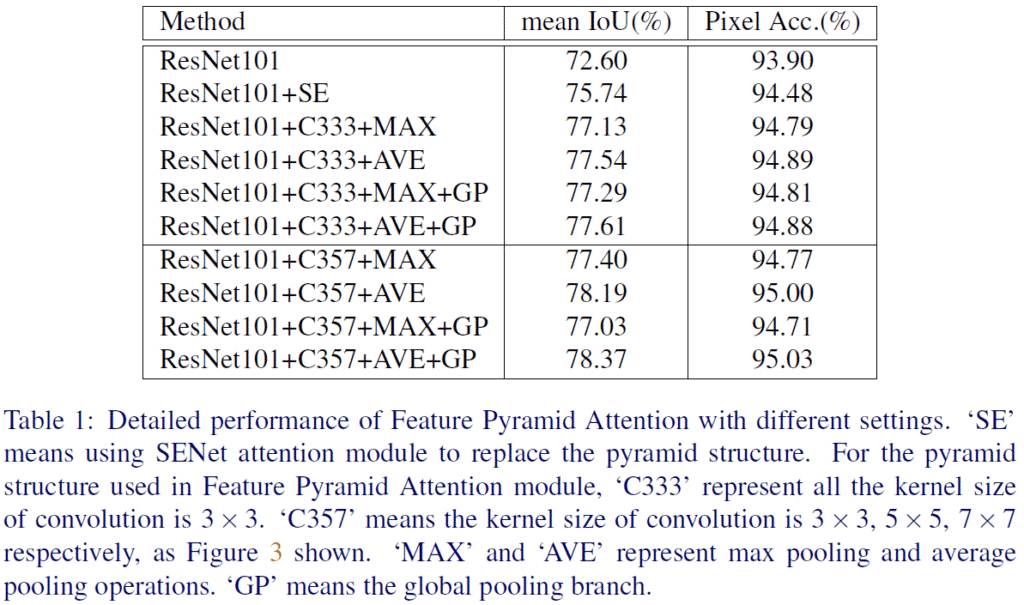
일반적으로 사용되는 아래 CE Loss의 구현을 Softmax를 빼고 보면 Loss Term에 Negative Class에 대한 고려가 들어가있지 않다. (마지막 정리부에서 다루지만 사실 CE Loss와 Softmax를 분리해서 보는 생각이 잘못된 것 같다.) 이 의문에서 시작해서 답을 찾아갔던 과정을 정리해보았다.
아래 수식에서 $p(x_{i})$는 정답값 분포 [0.0, 0.0, 1.0, 0.0, 0.0]를, $\hat{p}(x_{i})$는 Softmax를 거쳐나온 각 클래스별 확률 분포 $0<\hat{p}(x_{i})<1$를 의미한다.
$$-\sum_{i=1}^{C}{\{p(x_{i}){\cdot}log(\hat{p}(x_{i}))\}}$$
import numpy as np
# log(x) where 0 <= x < 1
# will return NaN or Inf if x == 0!
# We then add epsilon to x term.
eps = 1e-9
# Pseudo-outputs of softmax: softmax(logits)
# We can also calculate `logits` by applying
# inverse of the softmax: np.log(outputs)!
outputs = np.array([0.03, 0.05, 0.8, 0.1, 0.02])
label = np.array([0.0, 0.0, 1.0, 0.0, 0.0 ])
# CE Loss calculation
ce_loss = -np.sum(label * np.log(outputs + eps))
# > ce_loss: 0.22314355006420974CE Loss의 구현부를 보면 -np.sum(label * np.log(outputs + eps))으로 구성되는데, Negative Class의 Term은 label과 곱해지면서 상쇄되어 최종 Loss에 기여하지 않는다. 그러면 Softmax가 일반적으로 객체 분류문제에서 잘 작동하는 이유는 무엇일까? 내가 내린 결론은 Backpropagation에 있었다.
먼저, Stochastic Gradient Descent(SGD) 기법에서는 모델의 학습을 위한 방법으로 모델의 입력값에 대한 모델 가중치의 미분값Gradient을 구한다. 이를 더 쉽게 구하기 위하여 Chain Rule을 이용하며, 모델의 출력부부터 시작하여 모델의 입력부에 이르기까지 각 레이어별로 미분값을 구한다.
CE Loss $\mathcal{L}(s, y)$[1]만 고려했을 때, 이 Loss 함수를 입력값 $z$[2]에 미분하게 되면 다음과 같다. 상세한 미분 과정은 towardsdatascience 블로그(링크)에서 자세히 설명해주셨다.
$${{\partial{\mathcal{L}}}\over{\partial{\pmb{z}}}}=\pmb{s}-\pmb{y}$$
결국 Softmax 출력값과 레이블 값의 차이가 미분값이었던 것이다. 이렇게 함으로써 역전파Backpropagation 과정에서는 정답값이 아닌 클래스(0, 1, 3, 4번 클래스)의 확률도 잘 반영되어 학습이 진행되는 것이다.
Q. 그러면 CE Loss 값에는 Negative Class 값이 반영되지 않는가?
CE Loss는 Softmax 함수를 거친 뒤에 사용한다. 이를 중점으로 생각해 보면, Negative Class(0, 1, 3, 4번 Class)의 Logitsbefore softmax 값이 Positive Class(2번 Class)의 Probabilityafter softmax에 영향을 준다. 예를 들어, 아래 예시에서는 Negative Class의 영향력이 커질수록 Positive Class의 Probability는 작아진다.
import numpy as np
eps = 1e-9
# Define softmax (not used earlier)
def softmax(inputs: np.ndarray) -> np.ndarray:
exps = np.exp(inputs)
sums = np.sum(exps, axis=-1, keepdims=True)
return exps / sums
# Pseudo-outputs of softmax: softmax(logits)
# We can also calculate `logits` by applying
# inverse of the softmax: np.log(outputs)!
outputs = np.array([0.03, 0.05, 0.8, 0.1, 0.02])
label = np.array([0.0, 0.0, 1.0, 0.0, 0.0 ])
# Add arbitary value to logits and re-apply softmax
logits = np.log(outputs)
logits[0:2] += 0.4
logits[3:5] += 0.3
outputs_m = softmax(logits)
# Show differences between `outputs` and `outputs_m`
print(outputs_m)
print(outputs_m - outputs)
# > outputs_m:
# [0.04138864 0.06898107 0.73983032 0.12483331 0.02496666]
# > outputs_m - outputs:
# [0.01138864 0.01898107 -0.06016968 0.02483331 0.00496666]
# CE Loss calculation
ce_loss = -np.sum(label * np.log(outputs + eps))
# > ce_loss: 0.22314355006420974
ce_loss_m = -np.sum(label * np.log(outputs_m + eps))
# > ce_loss_m: 0.30133442040075237결국 CE Loss의 정의에서 input feature로는 probabilityoutput of softmax가 아니라, logitsbefore softmax를 중점으로 보았어야 했던 것이었다.
Conclusion
생각을 정리하다 보니, CE Loss와 Softmax를 한 묶음으로 생각하니까 모든 의문이 해결되었다. 처음에는 CE Loss term만 따로 두고 봐서 생겼던 의문이었지만, 결국 CE Loss의 정의에서 input feature로는 probabilityoutput of softmax가 아니라, logitsbefore softmax를 중점으로 보았어야 했던 것이었다.
본래 이 의문이 생겼던 것은, CE loss의 input probability를 변경하다가, 다른 값들을 넣어도 같은 loss value가 나왔던 것이 계기였다. 명백하게 확률의 분포가 다른데도 불구하고 loss 값이 동일하게 나와서 이에 의문이 들었었다.
outputs_1 = [0.05, 0.03, 0.8, 0.02, 0.1]
outputs_1 = [0.0, 0.0, 0.8, 0.2, 0.0]다시금 생각해보면 CE loss를 계산할 때 이러한 분포의 차이는 중요하지 않았다. label이 두 개 이상이라면 다르게 판단해야 할지도 모르겠지만, 하나인 이상 다른 클래스의 확률이 얼마나 큰 지는 중요하지 않은 것이다.
- [1] $s$: outputs of softmax, $y$: one hot vector of label
- [2] $z$: input logits of softmax



![Part Ⅶ. Semantic Segmentation] 6. DeepLab [1] - 라온피플 머신러닝 아카데미 - : 네이버 블로그](https://mblogthumb-phinf.pstatic.net/MjAxNzA1MDhfMTgg/MDAxNDk0MjA0NzM4MDA5._TkcVvaXD5-r-jyXciSTN-hKi1dga-Id3sNLDH3qpXIg.irXD5TPtE9L3UTi2jjgkgm9s6fkjx9xtbmCpiFSfg48g.PNG.laonple/%EC%9D%B4%EB%AF%B8%EC%A7%80_105.png?type=w2)